教育背景与工作经历
2022.07至今,bet体育365官网正规,副教授
2015.03-2022.07,香港科技大学生命科学部(张明杰院士课题组),博士后
2009.09-2014.09,厦门大学生命科学学院(林天伟教授课题组),博士
2005.09-2009.07,厦门大学生命科学学院,学士
研究兴趣
神经细胞突触可塑性的分子基础及其在学习与记忆中的作用
神经突触支架蛋白和信号蛋白的相互作用的结构基础
神经退行性疾病的致病机理
主要文章 (#: 共同第一作者)
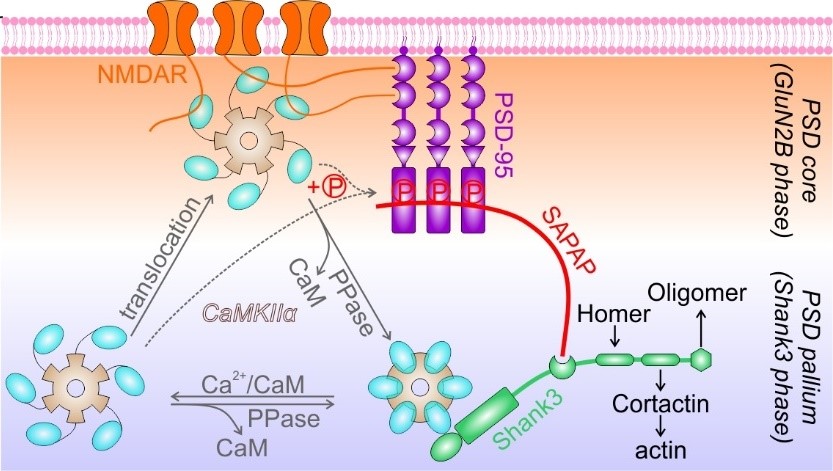
1. Cai, Q., Zeng, M., Wu, X., Wu, H., Zhan, Y., Tian, R., and Zhang, M. (2021) CaMKIIα-driven, phosphatase-checked postsynaptic plasticity via phase separation. Cell Res 31: 37-51.
CaMKIIα通过对钙信号的解码对神经突触的可塑性和学习起到重要作用。该工作证明了在钙信号和磷酸酶的调控下,CaMKIIα在不同条件下分别与Shank3和GluN2B相互作用。CaMKIIα在突触后致密区的不同区间的穿梭能够调控相分离介导的突触后库致密区的组装和突触可塑性。(封面文章)
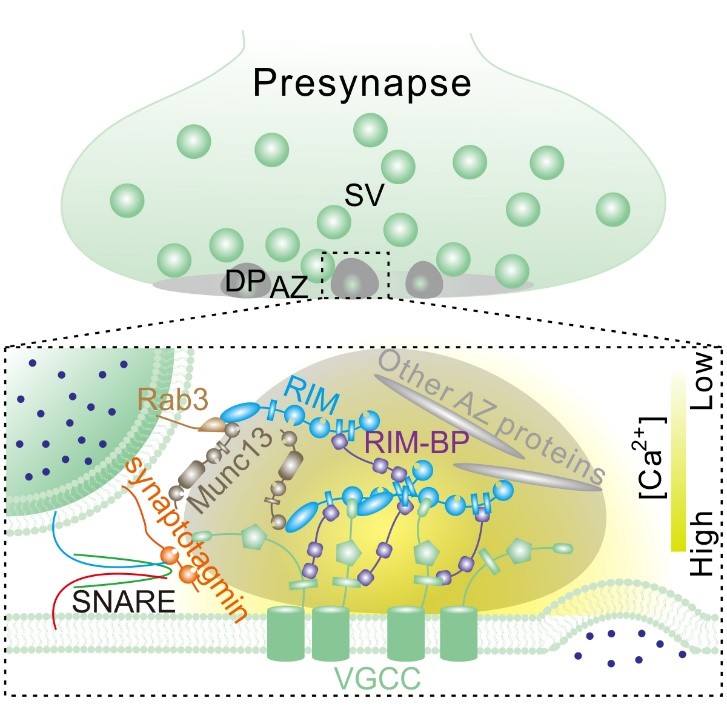
2. Wu, X.#, Cai, Q.#, Shen, Z., Chen, X., Zeng, M., Du, S. and Zhang, M. (2019) RIM and RIM-BP form presynaptic active-zone-like condensates via phase separation. Mol Cell 73 (5): 871-984.
神经递质释放的动力学取决于突触前膜上钙通道的富集和定位。该工作证明了突触前活性区的RIM和RIM-BP可以通过相分离形成动态且致密的组装。电压门控钙通道能被富集到RIM/RIM-BP的致密区中,从而帮助神经递质准确和高效率的释放。
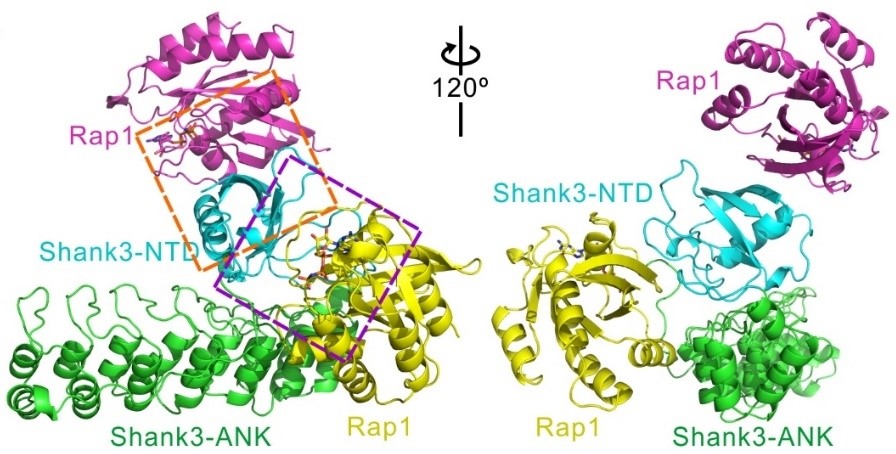
3. Cai, Q., Hosokawa, T., Zeng, M., Hayashi, Y. and Zhang, M. (2020) Shank3 binds to and stabilizes the active form of Rap1 and HRas GTPases via its NTD-ANK tandem with distinct mechanisms. Structure 28 (3): 290-300.
此工作解析了突触后致密区的主要支架蛋白Shank3的NTD-ANK结构域和Rap1以及HRas的复合物晶体结构,结合glutamate-uncaging实验揭示了Shank3与不同小G蛋白结合并把它们稳定在激活状态的不同机制。
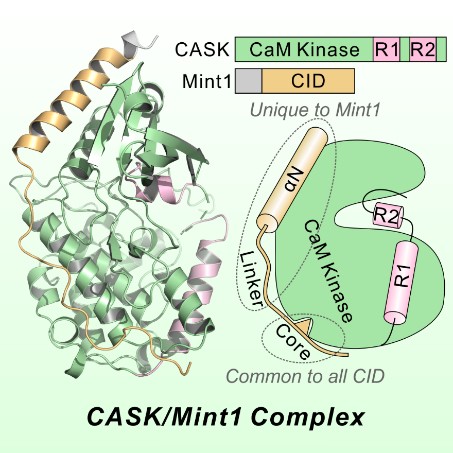
4. Wu, X.#, Cai, Q.#, Chen, Y., Zhu, S., Mi, J., Wang, J. and Zhang, M. (2020) Structural basis for the high-affinity interaction between CASK and Mint1. Structure 28 (6): 664-673.
此工作解析了突触前的支架蛋白CASK和Mint的复合物晶体结构,揭示了二者间强相互作用的分子基础。
5. Geng, Y.#, Liu, C.#, Cai, Q.#, Luo, Z., Miao, H., Shi, X., Xu, N., Fung, C.P., Choy, T.T., Yan, B., Li, N., Qian, P., Zhou, B., Zhu, G. (2021) Crystal structure of parallel G-quadruplex formed by the two-repeat ALS and FTD related GGGGCC sequence. Nucleic Acids Res 49 (10): 5881-5890.
6. Cai, Q.#, Yameen, M.#, Liu, W.#, Gao, Z., Li, Y., Peng, X., Cai, Y., Wu, C., Zheng, Q., Li, J. and Lin, T. (2013) Conformational plasticity of the 2A proteinase from enterovirus 71. J Virol 87: 7348-7356.
7. Wu, C.#, Cai, Q.#, Chen, C.#, Li, N., Peng, X., Cai, Y., Yin, K., Chen, X., Wang, X., Zhang, R., Liu, L., Chen, S., Li, J. and Lin, T. (2013) Structures of enterovirus 71 3C proteinase (strain E2004104-TW-CDC) and its complex with rupintrivir. Acta Crystallogr D Biol Crystallogr 69: 866-871.
8. Zhang, L.#, Huang, G.#, Cai, Q.#, Zhao, C., Tang, L., Ren, H., Li, P., Li, N., Huang, J., Chen, X., Guan, Y., You, H., Chen, S., Li, J., Lin, T. (2016) Optimize the interactions at S4 with efficient inhibitors targeting 3C proteinase from enterovirus 71. J Mol Recognit 29: 520-527.
9. Wu, C.#, Zhang, L.#, Li, P.#, Cai, Q.#, Peng, X., Yin, K., Chen, X., Ren, H., Zhong, S., Weng, Y., Guan, Y., Chen, S., Wu, J., Li, J., Lin, T. (2016) Fragment-wise design of inhibitors to 3C proteinase from enterovirus 71. BBA-Gen Subjects 1860: 1299-1307.